ความแตกต่างระหว่าง NGS กับ Sanger Sequencing | NGS vs Sanger Sequencing
ความแตกต่างที่สำคัญ - NGS และแซงเจอร์ลำดับ
ลำดับต่อเนื่อง (NGS) และแซงเจอร์ลำดับเป็นเทคนิคการจัดลำดับแบบ nucleotide 2 แบบที่พัฒนาขึ้นในช่วงเวลาดังกล่าว วิธีแซงเจอร์แซงเจอร์ใช้กันมาเป็นเวลาหลายปีแล้วและ NGS ได้เปลี่ยนมาใช้เมื่อเร็ว ๆ นี้เนื่องจากข้อดีของมัน ข้อแตกต่างที่สำคัญระหว่าง NGS และ Sanger Sequencing คือ NGS ทำงานบนหลักการ sequencing ล้าน sequences พร้อมกันอย่างรวดเร็วผ่านทางระบบ sequencing ในขณะที่ Sanger Sequencing ทำงานบนหลักของการสิ้นสุดโซ่เนื่องจากการเลือกใช้ของ dideoxynucleotides โดย เอนไซม์ดีเอ็นเอโพลาไรเซชันในระหว่างการจำลองแบบดีเอ็นเอและทำให้เกิดการแยกส่วนโดยการอิเล็กโทรฟิเรชั่นของเส้นเลือดฝอย
เนื้อหา
1 ภาพรวมและข้อแตกต่างที่สำคัญ
2. ลำดับนิวคลีโอไทด์คืออะไร
3. NGS คืออะไร
4. อะไรคือลำดับของแซงเจอร์
5. การเปรียบเทียบแบบเคียงข้างกัน - NGS vs. Sanger Sequencing
6. สรุป
ลำดับลำดับ nucleotide คืออะไร?
ข้อมูลทางพันธุกรรมถูกเก็บไว้ในลำดับเบสของดีเอ็นเอหรืออาร์เอ็นเอของเชื้อโรค กระบวนการในการกำหนดลำดับที่ถูกต้องของนิวคลีโอไทด์ (ใช้สี่ฐาน) ในส่วนที่กำหนด (ในยีนกลุ่มของยีนโครโมโซมและจีโนมสมบูรณ์) เรียกว่าลำดับเบส เป็นสิ่งที่สำคัญมากในการศึกษาทางพันธุกรรมการศึกษาทางนิติวิทยาศาสตร์ไวรัสวิทยาระบบชีวภาพการวินิจฉัยทางการแพทย์เทคโนโลยีชีวภาพและในสาขาอื่น ๆ เพื่อวิเคราะห์โครงสร้างและหน้าที่ของยีน มีวิธีการเรียงลำดับต่างๆที่พัฒนาขึ้นโดยนักวิทยาศาสตร์ ในหมู่พวกเขา Sanger sequencing ซึ่งพัฒนาโดย Frederick Sanger ในปี 2520 ได้รับการใช้กันอย่างแพร่หลายและเป็นที่นิยมมาเป็นเวลานานจนกว่าจะมีการแทนที่ Next Generation Sequencing
NGS คืออะไร?
ลำดับต่อไป (NGS) เป็นคำที่ใช้เรียกกระบวนการประมวลผลลำดับขั้นสูงที่ทันสมัย อธิบายถึงเทคโนโลยีการเรียงลำดับพันธุกรรมสมัยใหม่ที่แตกต่างกันซึ่งปฏิวัติการศึกษาทางพันธุกรรมและชีววิทยาโมเลกุล เทคนิคเหล่านี้ ได้แก่ การเรียงลำดับ Illumina การเรียงลำดับ Roche 454 การเรียงลำดับไอออนโปรตอนและการเรียงลำดับแบบลัด (sequencing by Oligo Ligation Detection) ระบบ NGS รวดเร็วและราคาถูกกว่า มีการใช้วิธีการเรียงลำดับดีเอ็นเอหลัก 4 วิธีในระบบ NGS คือ การเรียงลำดับตามลำดับโดยการสังเคราะห์การเรียงลำดับโดย ligation และการเรียงลำดับเซมิคอนดักเตอร์ไอออน จำนวนดีเอ็นเอหรืออาร์เอ็นเอเส้น (ล้าน) สามารถเรียงลำดับตามลำดับขนาน จะช่วยให้ลำดับของจีโนมทั้งหมดของสิ่งมีชีวิตภายในระยะเวลาอันสั้นซึ่งแตกต่างจากลำดับแซงเจอร์ซึ่งใช้เวลามากขึ้น
NGS มีข้อดีกว่าวิธีการเรียงลำดับทั่วไปของแซนเจอร์ เป็นกระบวนการที่มีความเร็วสูงถูกต้องและมีประสิทธิภาพมากขึ้นซึ่งสามารถทำได้โดยมีขนาดตัวอย่างเล็ก ๆ NGS สามารถนำมาใช้ในการศึกษาเกี่ยวกับเมทาจิโอนิกในการตรวจหารูปแบบภายในจีโนมบุคคลเนื่องจากการแทรกและการลบ ฯลฯ และในการวิเคราะห์การแสดงออกของยีน

รูปที่ 1: พัฒนาการในลำดับ NGS
Sequencing คืออะไร?
Sanger Sequencing เป็นวิธีการเรียงลำดับที่พัฒนาขึ้นโดย Frederick Sanger และเพื่อนร่วมงานของเขาเมื่อปีพ. ศ. 2520 เพื่อหาลำดับเบสของดีเอ็นเอที่กำหนดไว้อย่างถูกต้อง มันยังเป็นที่รู้จักกันในนาม chain sequence sequence sequence หรือ Dideoxy sequencing หลักการทำงานของวิธีนี้คือการสิ้นสุดของการสังเคราะห์เส้นใยโดยการผสมผสานการคัดแยก dideoxynucleotides (ddNTPs) เช่น ddGTP, ddCTP, ddATP และ ddTTP โดย DNA polymerase ในระหว่างการจำลองแบบของดีเอ็นเอ nucleotides ปกติมีกลุ่ม OH 3 'สำหรับการก่อตัวของพันธะ phosphodiester ระหว่าง nucleotides ที่อยู่ติดกันเพื่อต่อการสร้างเส้น อย่างไรก็ตาม ddNTPs ไม่มีกลุ่ม OH 3 และไม่สามารถสร้างพันธะ phosphodiester ระหว่าง nucleotides ได้ ดังนั้นการยืดตัวของโซ่จะหยุดลง
ในวิธีนี้ DNA ที่มีเกลียวแบบเดียวจะถูกจัดเรียงลำดับทำหน้าที่เป็นแม่แบบสำหรับการสังเคราะห์ดีเอ็นเอ ในหลอดทดลอง ข้อกำหนดอื่น ๆ ได้แก่ primer oligonucleotide, สารตั้งต้น deoxynucleotide และเอนไซม์ DNA polymerase เมื่อขนาบข้างปลายของชิ้นส่วนเป้าหมายเป็นที่รู้จักกันไพรเมอร์สามารถออกแบบได้ง่ายสำหรับการจำลองแบบดีเอ็นเอ ปฏิกิริยาการสังเคราะห์ดีเอ็นเอแยกจากกันสี่ครั้งจะทำในสี่หลอดแยกกัน แต่ละหลอดมี ddNTPs แยกกันและข้อกำหนดอื่น ๆ จากนิวคลีโอไทด์โดยเฉพาะจะมีส่วนผสมของ dNTPs และ ddNTPs ในทำนองเดียวกันปฏิกิริยาสี่แยกจะดำเนินการในสี่หลอดที่มีสี่ส่วนผสม หลังจากมีปฏิกิริยาเสร็จสิ้นการตรวจจับชิ้นส่วนดีเอ็นเอและการแปลงรูปแบบชิ้นส่วนเป็นข้อมูลลำดับ เศษดีเอ็นเอที่เกิดขึ้นจะถูกทำให้เสียสมบัติทางความร้อนและคั่นด้วยเจลอิเล็กโตรโฟเรซิส ถ้าใช้นิวคลีโอไทด์กัมมันตภาพรังสีรูปแบบแถบใน polyacrylamide gel สามารถมองเห็นได้ด้วย autoradiography เมื่อวิธีนี้ใช้ dideoxynucleotides ที่ติดแท็กเรืองแสงคุณสามารถลดเจลอ่านและส่งผ่านลำแสงเลเซอร์เพื่อตรวจจับโดยเครื่องตรวจจับเรืองแสงได้ เพื่อหลีกเลี่ยงข้อผิดพลาดที่อาจเกิดขึ้นเมื่อลำดับถูกอ่านด้วยตาและป้อนด้วยตนเองลงในคอมพิวเตอร์วิธีนี้พัฒนาขึ้นในการใช้ตัวเรียงลำดับอัตโนมัติควบคู่กับคอมพิวเตอร์
นี่คือวิธีการที่ใช้ในการสร้างลำดับดีเอ็นเอจากโครงการจีโนมมนุษย์ วิธีนี้ยังคงใช้อยู่กับการแก้ไขขั้นสูงเนื่องจากจะให้ข้อมูลลำดับที่ถูกต้องแม้จะเป็นกระบวนการที่มีราคาแพงและช้าก็ตาม
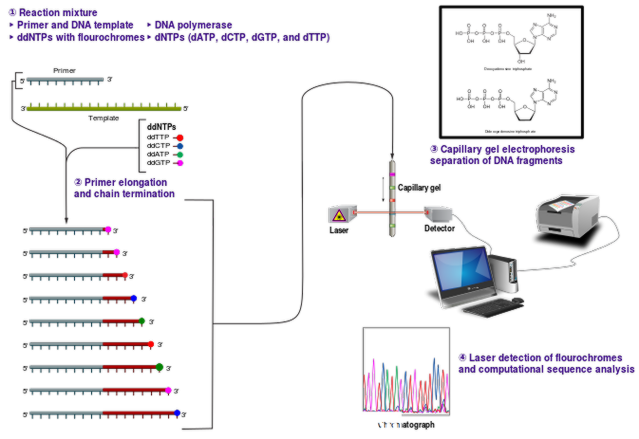
รูปที่ 2: Sanger Sequencing
อะไรคือข้อแตกต่างระหว่าง NGS กับ Sanger Sequencing?
- diff บทความ Middle ก่อน Table ->
NGS และ Sanger Sequencing |
|
| Sequencing ยุคถัดไป (NGS) หมายถึงกระบวนการจัดเรียงลำดับขั้นสูงที่ทันสมัยอธิบายลำดับเทคโนโลยีลำดับขั้นที่แตกต่างกันจำนวนมาก | Sanger Sequencing เป็นวิธีการเรียงลำดับที่พัฒนาขึ้นโดย Frederick Sanger เพื่อกำหนดลำดับ nucleotide ที่แม่นยำของส่วนดีเอ็นเอที่กำหนด |
| ต้นทุนประสิทธิผล | |
| NGS เป็นกระบวนการที่ถูกกว่าเพราะลดเวลาพลังงานของมนุษย์และสารเคมี | นี่เป็นกระบวนการที่ต้องเสียค่าใช้จ่ายเนื่องจากต้องใช้เวลาใช้กำลังคนและสารเคมีมากขึ้น |
| ความเร็ว | |
| เร็วขึ้นเนื่องจากการตรวจจับสารเคมีและการตรวจจับสัญญาณของเส้นใยหลาย ๆ คู่เกิดขึ้นแบบคู่ขนาน | ใช้เวลานานนับตั้งแต่การตรวจจับสารเคมีและการตรวจจับสัญญาณเกิดขึ้นเป็นสองกระบวนการแยกกันและเฉพาะในเส้นใยสามารถอ่านได้ในแต่ละครั้ง |
| ความน่าเชื่อถือ | |
| NGS มีความน่าเชื่อถือ | ลำดับแซนเจอร์มีความน่าเชื่อถือน้อย |
| ขนาดตัวอย่าง | |
| NGS ต้องใช้ดีเอ็นเอจำนวนน้อย | วิธีนี้ต้องการ DNA เทมเพลตจำนวนมาก |
| ฐาน DNA ต่อชิ้นส่วนที่เรียงตามลำดับ | |
| จำนวนฐาน DNA ที่มีต่อลำดับเบสต่ำกว่าวิธีแซงเจอร์ | ลำดับการสร้างมีความยาวมากกว่าลำดับ NGS |
สรุป - NGS และ Sanger Sequencing
NGS และ Sanger Sequencing เป็นเทคนิคการหาลำดับเบสที่ใช้กันอย่างกว้างขวางในชีววิทยาโมเลกุล ลำดับแซนเจอร์เป็นวิธีเรียงลำดับแรกซึ่งถูกแทนที่โดย NGS ความแตกต่างหลักระหว่าง NGS และแซงเจอร์ลำดับคือ NGS เป็นกระบวนการที่มีความเร็วสูงถูกต้องและคุ้มค่ากว่าการเรียงลำดับแซนเจอร์ เทคนิคทั้งสองนี้ก่อให้เกิดการระบาดครั้งใหญ่ในพันธุศาสตร์และเทคโนโลยีชีวภาพ
การอ้างอิง:
1. Nowrousian, Minou เทคนิคการจัดลำดับถัดไปของจุลินทรีย์ที่มียูคาริโอต: วิธีการแก้ปัญหาทางชีวภาพแบบลำดับต่อเนื่อง เซลล์ Eukaryotic สมาคมจุลชีววิทยาอเมริกัน, กันยายน 2553 18 ก.พ. 2017
2. Sanger, F., S. Nicklen และ A. R. Coulson "การจัดลำดับดีเอ็นเอด้วยสารยับยั้งโซ่ - ปลาย "การดำเนินการของสถาบันวิทยาศาสตร์แห่งชาติ 74. 12 (1977): 5463-467. เว็บ.
3 Liu, Lin, Yinhu Li, Siliang Li, Ni Hu, Yimin He, Ray Pong, Danni Lin, Lihua Lu และ Maggie Law "เปรียบเทียบระบบ sequencing ของยุคหน้า "วารสารชีวศาสตร์และเทคโนโลยีชีวภาพ 2012 (2012): 1-11. เว็บ.
Image Courtesy:
"Sanger-sequencing" โดย Estevezj - งานของตัวเอง (CC BY-SA 3. 0) ผ่านทาง Commons Wikimedia
"การพัฒนาลำดับถัดไป" โดย Nederbragt, Lex (2012) - (CC BY 3. 0) ผ่านวิกิพีเดีย:


