การซ่อมแซมที่ไม่ตรงกันและการตัดไม้ทำลายสารนิวคลีโอไทด์
ความแตกต่างที่สำคัญ - การซ่อมแซมที่ไม่ตรงกันและการกำจัดสารเคมีด้วยความผิดพลาดของ Nucleotide
นับสิบพันของความเสียหายของ DNA เกิดขึ้นในเซลล์ต่อวัน ก่อให้เกิดการเปลี่ยนแปลงในกระบวนการของเซลล์เช่นการจำลองแบบการถอดรหัสรวมทั้งความสามารถในการทำงานของเซลล์ ในบางกรณีการกลายพันธุ์ที่เกิดจากความเสียหายของดีเอ็นเอเหล่านี้อาจทำให้เกิดโรคที่เป็นอันตรายเช่นโรคมะเร็งและโรคที่เกี่ยวข้องกับอายุ (เช่น Progeria) โดยไม่คำนึงถึงความเสียหายเหล่านี้เซลล์เริ่มต้นกลไกการซ่อมแซมน้ำตกที่มีการจัดระเบียบอย่างมากซึ่งเรียกว่าการตอบสนองต่อความเสียหายของดีเอ็นเอ มีการระบุระบบซ่อม DNA หลายระบบในระบบเซลลูล่าร์ (BER), การซ่อมแซมที่ไม่ตรงกัน (MMR), การซ่อมแซมการตัดใหม่ของ Nucleotide (NER), การซ่อมแซมชิ้นส่วนแบบ Double Strand การซ่อมแซมการตัดตอนนิวเคลียสเป็นระบบอเนกประสงค์ที่สามารถรับรู้ถึงความผิดเพี้ยนของแผลเลียนแบบเกลียวของเกลื้อนขนาดใหญ่และกำจัดออกได้ ในทางตรงกันข้ามการซ่อมแซมที่ไม่ตรงกันจะแทนที่ฐานข้อมูล misincorporated ระหว่างการจำลองแบบ ข้อแตกต่างที่สำคัญระหว่างการซ่อมแซมที่ไม่ตรงกันและการซ่อมแซมการตัดใหม่ของนิวคลีโอไทด์คือการซ่อมแซมการตัดใหม่ของนิวคลีโอไทด์ (NER) เพื่อใช้ในการกำจัด dimer pyrimidine ที่เกิดจากการฉายรังสี UV และแผลเป็นเกลื้อนขนาดใหญ่ที่เกิดจากสารเคมีในขณะที่ระบบซ่อมแซมไม่ตรงกันมีบทบาทสำคัญในการแก้ไข misincorporated ฐานที่หลบหนีจากเอนไซม์การจำลองแบบ (DNA polymerase 1) ในระหว่างการทำซ้ำ นอกเหนือไปจากฐานที่ไม่ตรงกันแล้วโปรตีนในระบบ MMR ยังสามารถซ่อมแซมส่วนแทรก / ลบลูป (IDL) ซึ่งเป็นผลของความลื่นไถลของโพลิเมอร์ในระหว่างการทำสำเนาดีเอ็นเอซ้ำ ๆ
เนื้อหา
1 ภาพรวมและข้อแตกต่างที่สำคัญ
2. ซ่อมไม่ตรงกันคืออะไร
3. การกำจัดสารนิวคลีโอไทด์คืออะไร
4. การเปรียบเทียบแบบเคียงข้างกัน - การซ่อมแซมที่ไม่ตรงกันและการตัดไม้ทำลายด้วยนิวเคลียส
5. สรุป
การซ่อมแซมสารนิวคลีโอไทด์คืออะไร?
คุณลักษณะที่โดดเด่นที่สุดของการซ่อมแซมการตัดตอนนิวคลีโอไทด์คือการซ่อมแซมความเสียหายที่เกิดจาก nucleotide ที่เกิดจากการบิดเบือนอย่างมากในเกลียวคู่ของดีเอ็นเอ เป็นที่สังเกตในเกือบทุกสิ่งมีชีวิตที่ได้รับการตรวจสอบถึงวันที่ Uvr A, Uvr B, Uvr C (excinucleases) Uvr D (helicase) เป็นเอนไซม์ที่รู้จักกันดีที่สุดที่เกี่ยวข้องกับ NER ซึ่งกระตุ้นให้เกิดการซ่อมแซม DNA ในรูปแบบของ Ecoli คอมเพล็กซ์เอนไซม์ย่อยหลาย Uvr ABC ผลิต Uvr A, Uvr B, Uvr C polypeptidesยีนที่เข้ารหัสสำหรับ polypeptides ดังกล่าว ได้แก่ uvr A, uvr B, uvr C. เอนไซม์ Uvr A และ B สามารถรับรู้ความเสียหายที่เกิดจากความผิดเพี้ยนที่เกิดจากเกล็ดคู่แบบ DNA เช่น dimer pyrimidine เนื่องจากการฉายรังสีด้วยรังสียูวี Uvr A เป็นเอนไซม์ ATPase และนี่คือปฏิกิริยาการวิเคราะห์ด้วยตัวทำละลาย จากนั้น Uvr A จะออกจากดีเอ็นเอในขณะที่ Uvr BC complex (nuclease ที่ใช้งานอยู่) จะยึด DNA ไว้ในทั้งสองด้านของความเสียหายที่เร่งด้วย ATP โปรตีนอื่นที่เรียกว่า Uvr D ที่เข้ารหัสโดยยีน uvrD เป็นเอนไซม์ helicase II ช่วยปลดปล่อยดีเอ็นเออันเป็นผลมาจากการปลดปล่อยชิ้นส่วนดีเอ็นเอที่เสียหายเพียงครั้งเดียว นี่เป็นช่องว่างในเกลียวดีเอ็นเอ หลังจากแบ่งส่วนที่ได้รับความเสียหายแล้วจะมีช่องว่าง DNA 12-13 ช่องโหว่อยู่ นี่คือเติมด้วยดีเอ็นเอโพลิเมอร์เอนไซม์ I และกรงขังผนึกด้วยดีเอ็นเอ ligase ต้องใช้ ATP ในขั้นตอนที่สามของปฏิกิริยานี้ กลไก NER สามารถระบุได้ในมนุษย์ที่มีสัตว์เลี้ยงลูกด้วยนมเช่นกัน ในมนุษย์สภาพผิวที่เรียกว่า Xeroderma pigmentosum เป็นสาเหตุมาจากดีเอ็นเอ dimers ที่เกิดจากการฉายรังสียูวี ยีน XPA, XPB, XPC, XPD, XPE, XPF และ XPG ผลิตโปรตีนเพื่อทดแทนความเสียหายของดีเอ็นเอ โปรตีนของยีน XPA, XPC, XPE, XPF และ XPG มีกิจกรรม nuclease ในทางกลับกันโปรตีนของยีน XPB และ XPD แสดงกิจกรรม helicase ซึ่งทำหน้าที่คล้ายคลึงกับ Uvr D ใน E coli

รูปที่ 01: การซ่อมแซมการตัดไม้ทำลายนิวเคลียร์
Mismatch Repair คืออะไร?
ระบบการซ่อมแซมที่ไม่ตรงกันเกิดขึ้นระหว่างการสังเคราะห์ดีเอ็นเอ แม้จะมียูนิตย่อยที่ทำงานได้แล้วก็ตามดีเอ็นเอโพลิเมอร์ III สามารถรวม nucleotide ผิด ๆ สำหรับการสังเคราะห์ได้ทุกๆ 10 8 คู่เบส โปรตีนซ่อมแซมที่ไม่ตรงกันจะจดจำ nucleotide นี้ได้และแทนที่ด้วย nucleotide ที่ถูกต้องสำหรับระดับความถูกต้องสุดท้าย การสร้างเมธิลดีเอ็นเอเป็นส่วนสำคัญสำหรับโปรตีน MMR ในการรับรู้เส้นใยแม่จากเส้นใยสังเคราะห์ใหม่ methylation ของ adenine (A) nucleotide ใน motif GATC ของเส้นใยสังเคราะห์ใหม่จะล่าช้าเล็กน้อย ในทางตรงกันข้ามกลุ่มแม่แบบ adenine nucleotide ในแม่ลายของ GATC ได้ทำ methylated ไว้แล้ว โปรตีน MMR รู้จักเส้นสังเคราะห์ใหม่โดยความแตกต่างนี้จากเส้นแม่และเริ่มต้นการซ่อมแซมที่ไม่ตรงกันในเส้นใยสังเคราะห์ใหม่ก่อนที่มันจะกลายเป็น methylated โปรตีน MMR กำกับกิจกรรมการซ่อมแซมของพวกเขาเพื่อลดนิวคลีโอไทด์ผิดก่อนที่ดีเอ็นเอที่จำลองใหม่จะได้รับ methylated เอนไซม์ Mut H, Mut L และ Mut S ที่เข้ารหัสโดยยีน mut H, mut L, mut S กระตุ้นปฏิกิริยาเหล่านี้ใน Ecoli โปรตีน Mut S รู้จัก 7 คู่ของคู่เบสที่ไม่สามารถจับคู่ได้ถึง 7 คู่ยกเว้น C: C และผูกไว้ที่ตำแหน่งที่ไม่ตรงกันในดีเอ็นเอดูเพล็กซ์ ด้วย ATP ที่ถูกผูกไว้ Mut L และ Mut S เข้าร่วมในภายหลัง ที่ซับซ้อน translocates ไม่กี่พันคู่ฐานออกไปจนกว่าจะพบ motif hemimethylated GATC กิจกรรมของ nuclease ที่หยุดนิ่งของโปรตีน Mut H จะเริ่มทำงานเมื่อพบ motif hemimethylated GATC ตัดเส้นใยดีเอ็นเอที่ไม่ได้เมมเลตออกจากนิเกิล 5 'ที่แกนกลางของโมเลกุล GATC ที่ไม่ได้เลียนแบบ (เส้นใยดีเอ็นเอสังเคราะห์ใหม่)ในส่วนที่เหลือการกระทำโดยรวมของ Uvr D เป็นโปรตีน helicase, Mut U, SSB และ exonuclease ฉันจะยกเว้นค่า nucleotide ที่ไม่ถูกต้องใน single-stranded ดีเอ็นเอ. ช่องว่างที่เกิดขึ้นในการตัดตอนจะเต็มไปด้วย DNA polymerase III และปิดด้วย ligase ระบบเดียวกันนี้สามารถระบุได้ในหนูและมนุษย์ การกลายพันธุ์ของมนุษย์ hMLH1, hMSH1 และ hMSH2 มีส่วนเกี่ยวข้องกับมะเร็งลำไส้ใหญ่ nonpolyposis ทางพันธุกรรมซึ่งทำให้เซลล์แบ่งเซลล์ออกเป็นสองส่วน
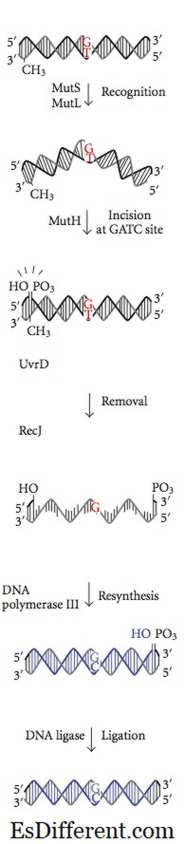
- diff บทความกลางก่อนตาราง ->
การซ่อมแซมที่ไม่ตรงกันและการซ่อมแซมการตัดไม้ทำลายนิวเคลียส
ระบบซ่อมแซมที่ไม่ตรงกันเกิดขึ้นในระหว่างการจำลองแบบโพสต์
เกี่ยวข้องกับการขจัด dimer pyrimidine เนื่องจากการฉายรังสียูวีและแผลของดีเอ็นเออื่น ๆ เนื่องจากสารตัวเติมเคมี |
|
| เอนไซม์ | เป็นสารเร่งปฏิกิริยาโดย Mut S, Mut L, Mut H, Uvr D, SSB และ Exonuclease I. |
| เป็นตัวเร่งปฏิกิริยาด้วย Uvr A, Uvr B, Uvr C, UvrD เอนไซม์ | |
| Methylation | เป็นหัวใจสำคัญในการเริ่มต้นปฏิกิริยา |
| การสร้างเมทิลเลตดีเอ็นเอไม่จำเป็นสำหรับการเริ่มต้นปฏิกิริยา | |
| การกระทำของเอนไซม์ | Mut H คือ endonuclease. |
| Uvr B และ Uvr C เป็น exonucleases | |
| โอกาส | เหตุการณ์นี้เกิดขึ้นเฉพาะระหว่างการจำลองแบบ |
| สิ่งนี้เกิดขึ้นเมื่อสัมผัสกับ U. V หรือสารเคมีที่กลายพันธุ์ไม่อยู่ระหว่างการทำซ้ำ | |
| การอนุรักษ์ | มันมีการอนุรักษ์สูง |
| มันไม่ได้มีการอนุรักษ์อย่างมาก | |
| Gap Filling | ทำโดย DNA polymerase III |
| ข้อมูลนี้เป็นคำอธิบายโดยย่อเกี่ยวกับ DNA polymerase I. | |
| สรุป - การซ่อมแซมที่ไม่ตรงกัน (Mismatch Repair vs Nucleotide Excision Repair) | การซ่อมแซมที่ไม่ตรงกัน (MMR) และการซ่อมแซมการตัดใหม่ของ Nucleotide (NER) เป็นกลไกสองอย่างที่เกิดขึ้นในเซลล์เพื่อแก้ไข ความเสียหายของดีเอ็นเอและความผิดเพี้ยนที่เกิดจากตัวแทนต่างๆ เหล่านี้รวมชื่อเป็นกลไกการซ่อมแซมดีเอ็นเอ การซ่อมแซมการตัดไม้ทำลายนิวเคลียสทำให้เกิดความเสียหายกับนิวคลีโอไทด์ที่เปลี่ยนแปลงไปซึ่งโดยปกติแล้วความเสียหายที่สำคัญเหล่านี้เกิดจากดีเอ็นเอเกลียวคู่ที่เกิดขึ้นเนื่องจากการสัมผัสกับการฉายรังสียูวีและสารเคมี โปรตีนซ่อมแซมที่ไม่ตรงกันจะจำแนกนิวคลีโอไทด์ผิด ๆ ยกเว้นสารเคมีและแทนที่ด้วยนิวคลีโอไทด์ที่ถูกต้อง ขั้นตอนนี้รับผิดชอบขั้นตอนสุดท้ายของความถูกต้องในระหว่างการจำลองแบบ |
การอ้างอิง:
1. Cooper, Geoffrey M. "การซ่อมแซมดีเอ็นเอ "เซลล์: วิธีการเชิงโมเลกุล ฉบับที่ 2 U. S. หอสมุดแห่งชาติแพทยศาสตร์, 01 มกราคม 1970. Web 09 มี.ค. 2017.
2. กลไกและหน้าที่ของการซ่อมแซมดีเอ็นเอที่ไม่ตรงกัน "การวิจัยเซลล์ U. S. หอสมุดแห่งชาติแพทยศาสตร์ n. d เว็บ. 09 มี.ค. 2017.
รูปภาพมารยาท:
1. "สมุดบันทึกการตัดไม้ทำลายล้างด้วย Nucleotide pbio 0040203. g001 "โดย Jill O. Fuss, Priscilla K. Cooper - (CC BY 2. 5) โดยวิกิมีเดีย
2. "ดีเอ็นเอไม่ตรงกันการซ่อมแซม Ecoli" โดย Kenji Fukui - (CC BY 4. 0) via Commons Wikimedia




